Whole genome sequencing data used for surveillance of Campylobacter infections: detection of a large continuous outbreak, Denmark, 2019
Eurosurveillance, Volume 26, Issue 22
03/Jun/2021
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8176674/pdf/eurosurv-26-22-1.pdf(論文PDF)
https://www.eurosurveillance.org/content/10.2807/1560-7917.ES.2021.26.22.2001396
(食品安全情報2021年24号(2021/11/24)収載)
背景
カンピロバクターは、細菌性胃腸疾患で最も高頻度に検出される病原体の1つである。カンピロバクター症アウトブレイクの報告はまれであるが、これは通常のサーベイランスにおいて分子生物学的タイピングが行われないためである可能性がある。患者由来カンピロバクター分離株の全ゲノムシークエンシング(WGS)解析データを使用すると、アウトブレイクに似た小規模なクラスターが多数検出されることが以前の報告で示されている。
目的
遺伝子型解析にもとづくカンピロバクター症サーベイランスは2019年に開始された。その目的は、患者由来分離株の大規模なクラスターの検出を可能にすること、および発生中のアウトブレイクに対応するためにアウトブレイク期間中に販売された鶏肉からの分離株と患者由来分離株との比較を行うことであった。
方法
2019年に、患者由来分離株(n=701)および鶏肉由来分離株(n=164)のWGS解析が行われた。患者由来分離株のクラスターの検出および患者由来分離株と鶏肉由来分離株との比較には、コアゲノム多座塩基配列タイピング(cgMLST)法が使用された。
結果
72のクラスターが検出され、内訳は小規模(患者数2〜4人)な58クラスターおよび大規模(患者数5〜91人)な14クラスターであった(表)。患者由来分離株の3分の1のタイピング結果が鶏肉由来分離株と一致した。
表:患者由来カンピロバクター属菌の全クラスターおよび鶏肉由来株とタイピング結果が一致する患者由来カンピロバクター属菌のクラスター(デンマーク、2018年10月〜2019年12月)
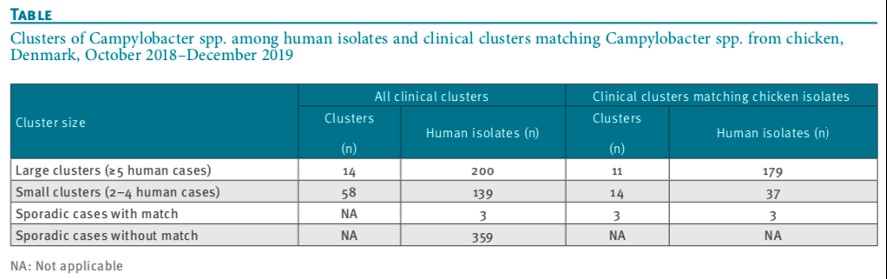
NA:データなし
大規模な1クラスター(塩基配列型(ST)122#1クラスター)が1年間を通じて存在しており、検査を行ったカンピロバクター症患者全員のうちの12%を占めた(図)。このクラスターのタイプに該当するカンピロバクターが食鳥処理場1カ所に由来する複数の鶏肉検体で認められ、当該食鳥処理場でアウトブレイク対策が実施された。
図:塩基配列解析を行った患者由来カンピロバクター分離株の塩基配列型(ST)122#1およびその他のSTの月別分離株数(デンマーク、2018年10月〜2019年12月、n=701)
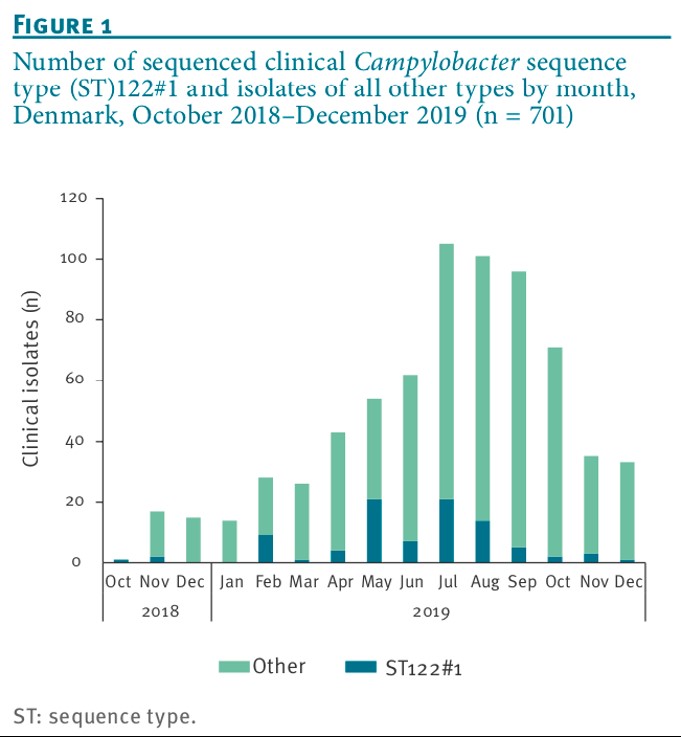
ST:塩基配列型
結論
WGS法を用いたサーベイランスは、鶏肉におけるカンピロバクター汚染の動態およびヒト患者クラスターとの関連を把握するのに有用であった。
国立医薬品食品衛生研究所 安全情報部